はじめに
これまで、さまざまな人工アミノ酸配列を、タンパク質としてモデリングして3Dプリンターに出力してきました。

活動の背景としては、
- タンパク質科学とアートのつながりを強めたい
- タンパク質の構造決定に関わる知見を深めたい
という2つの想いがあります。
しかし、前者にこだわりすぎると、デザインするタンパク質は複雑になっていきます。タンパク質はヘリックスやシート構造など普遍的な2次構造をとるため、短すぎる配列をモデリングしても、構造の多様性が乏しくなってしまうためです。
一方で、全長配列の長いタンパク質の構造は、様々な要素が組み合わさって一つの形をつくるため、その特徴を考察したり、まして普遍的な真理を導くことは非常に困難に感じました。
Foldit
Folditという、タンパク質の正しいフォールディングを予想するオンラインゲームがあります。ほどかれた構造を与えられたプレイヤーは、分子を部分的に引っ張ったり、回転させたりして構造を変化させることができます。動かすごとにその時点での安定化エネルギーを反映した値がリアルタイムで増減しますので、それを指標に安定な構造を探索し、高いスコアを競うゲームです。
本ゲームについては、後日別の記事でも解説しますので、ご期待ください。
可視化されたタンパク質構造を自由に動かし、その構造を評価することができるツールは、タンパク質フォールディングの仕組みを紐解くのに、非常に役立つと思いました。
しかしゲームのFolditは、任意のアミノ酸配列を触ることはできず、ゲーム側で提供されたタンパク質を扱うことに限定されます。またその配列も視認することも(おそらく)難しいです。
一方で、Foldit Standaloneと呼ばれる、ソフトウェアパッケージも存在します。こちらはオフラインでFolditの操作環境を扱えるツールです。ここではfastaファイル、またはPDBファイルをインポートすることで、自身の好きなタンパク質をFoldit上で扱うことができます。そこで、このツールを利用して最良のスコアを目指しタンパク質を操作することで、タンパク質構造解析の学習に役立てることができるか、検証してみることにしました。
触れてみた感想
Folditにインポートしてみた配列は、以前に記事で紹介したこちらのアミノ酸配列です。
アミノ酸配列:AHAPPYNEWYEAR
ひとまず、短めの分子を選びました。
最初に、fastaファイルを作成して、配列としてFolditに渡すと、こちらのようにほどかれた状態で構造が描画されます(図1)。

つぎに、Alphafoldで予測したPDBファイルをロードしてみます(図2)。

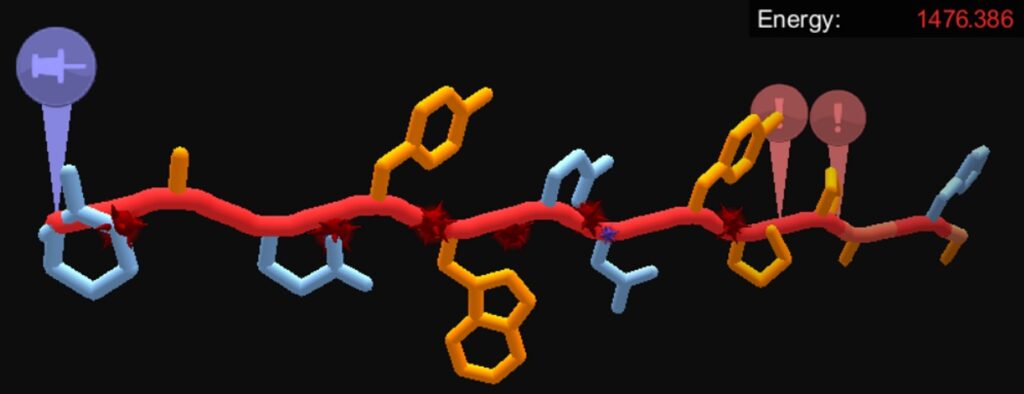
主鎖が複雑に折りたたまれて、スコアが高くなっていることがわかります。
図2の〇で囲った領域は、TrpとArgの側鎖が明らかにぶつかっていました。このクラッシュを解消しようと試みると、なぜかスコアが低くなりました(図3)。Trpの表面露出が望ましくないということでしょうか。このように直感的にクラッシュを取り除こうとすると、ほとんどのケースでスコアが低くなる傾向にありました。

主鎖中央のTyr残基にも、クラッシュが確認できます(図4)。取りうる角度が数通りに限定されているため、どの方向に向けてもいずれかの側鎖とクラッシュしてしまいます。

ちなみに、図1から、AFモデルを無視して最良のスコアを目指してみると、主鎖を2つに折りたたんだ構造が最も高いスコアを示しました(図5)。

終わりに
操作とスコアの増減の因果関係がとても難しいです。スコアが実態を正しく反映しているか確認したいので、構造既知のペプチドで試してみるのも良いかもしれません。Folditの操作が不慣れな面もあるので、ゲームで鍛えていきたいと思いました。



コメント